电话:027-87686090
地址:湖北省武汉市洪山区珞喻路237号 (430079)
邮箱:esev2022@126.com
高通量小RNA-seq技术为全面研究EV中的小RNA 提供了绝佳机会。迄今为止,很多数据库从细胞外囊泡中挑选了几种分子进行了注释分析。例如,Vesiclepedia、ExoCarta和EVpedia从已有的研究中挑选出来源于细胞外囊泡中的四种类型的生物活性分子(蛋白质、RNA、代谢物和脂质)。除此之外,一些数据库还从细胞外囊泡的测序数据中深入评估了 RNA 的丰度。目前,exRNA Atlas是最大的细胞外RNA(exRNA)表达图谱数据库,其中包含159个EV和205个MV样本。而 ExoRBase 专注于 87 个EV样本。ExoBCD对来自4 个数据集的乳腺癌中的差异表达基因和细胞外囊泡miRNA/lncRNA进行了分析。EVmiRNA是一个专注于EV中miRNA 表达谱的综合数据库。EVAtlas是EVmiRNA的升级版,与EVmiRNA 相比,增加了两倍的数据量以及7种类型的小RNAs,并且更多地关注EV中的ncRNA功能。由于这些细胞外囊泡相关的小RNA数据库对于研究细胞外囊泡的功能有重要作用,因此我们在这一节详细介绍几个比较有代表性的细胞外囊泡中转录组相关的数据库,如EVmiRNA和EVAtlas。除此之外,还列出了一些常见的细胞外囊泡的数据库,感兴趣的用户可以到对应的网站上进一步了解和使用。
EVmiRNA收集了细胞外囊泡中的miRNA表达谱。在EVmiRNA 数据库中,作者分析了来自 17种组织/疾病的细胞外囊泡来源的462个smRNA测序数据集(如图1)。EVmiRNA数据库中包含了miRNA 表达谱、miRNA调节通路、miRNA功能、miRNA相关药物以及相对应的文献。
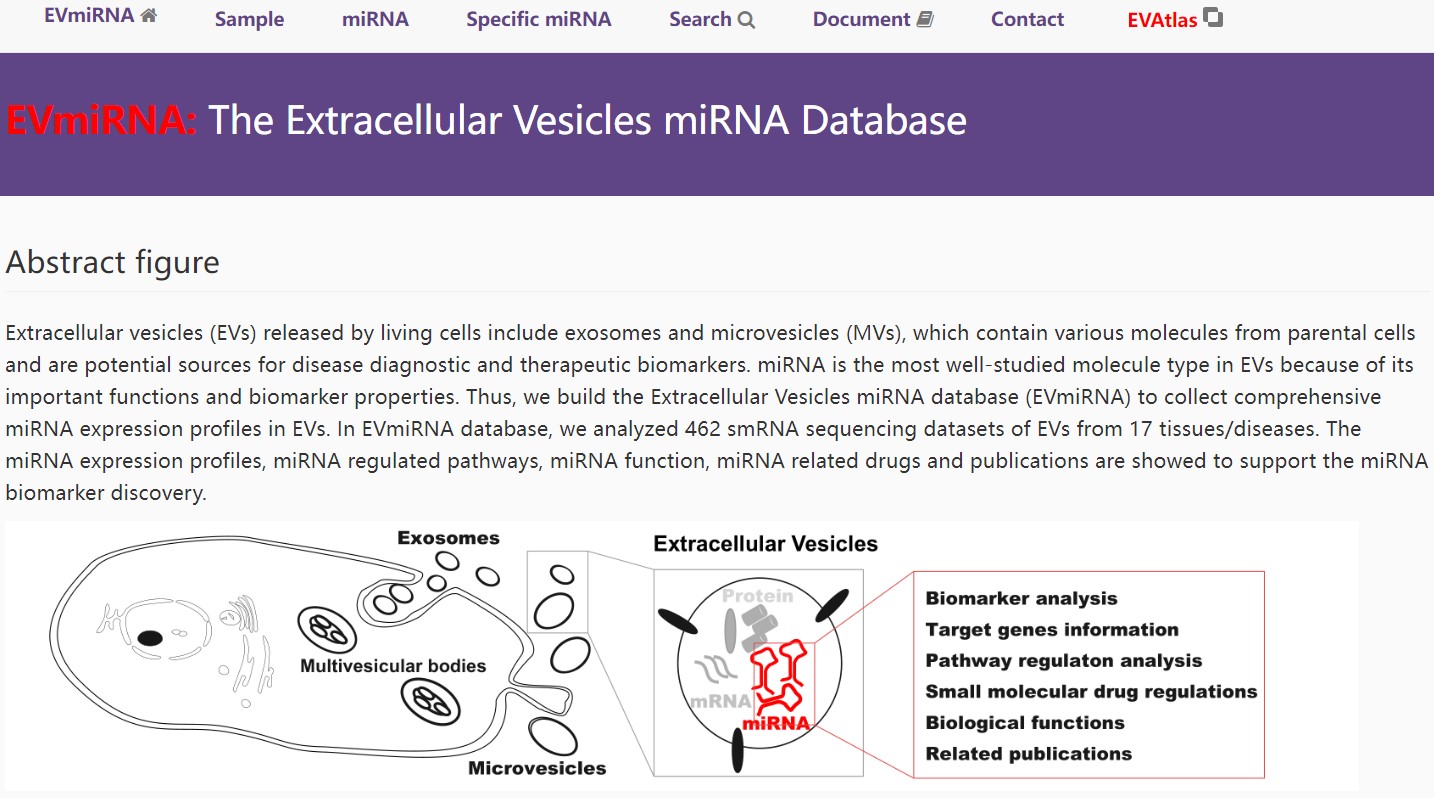
图1 EVmiRNA数据库首页
该数据库的使用十分简单(如图2),用户在首页的两个搜索栏中输入感兴趣的miRNA或者输入作者提供的样例,或者再在右边选择不同的EV来源等限制条件,如给出的“Blood-exosomes”,再点击搜索(“Search”)即可(图2A)。得到的结果中包含了7类信息,分别是“Basic information of miRNA”,“miRNA Expression”,“Pathway information”,“Function”,“Small molecular drug”,“Publication”以及“miRNA target information”(图2B)。除此之外,用户还可以根据顶部导航栏点击不同的模块快速浏览和查询细胞外囊泡中miRNA相关的信息(图2C)。

图2 EVmiRNA数据库的使用示例
EVAtlas是 EVmiRNA 数据库的更新。该数据库采用了一个名为动态分配的算法 (RDAA,英文全称),考虑了短序列在匹配到多种小RNA序列上如何分配的问题。最终量化了超过2030个样本中7种ncRNA类型(miRNA、snoRNA、piRNA、snRNA、rRNA、tRNA 和 Y RNA)的表达谱。这些样本来自24种不同条件(组织、疾病和正常)下的细胞外囊泡的小RNA-seq测序数据集(如图3)。
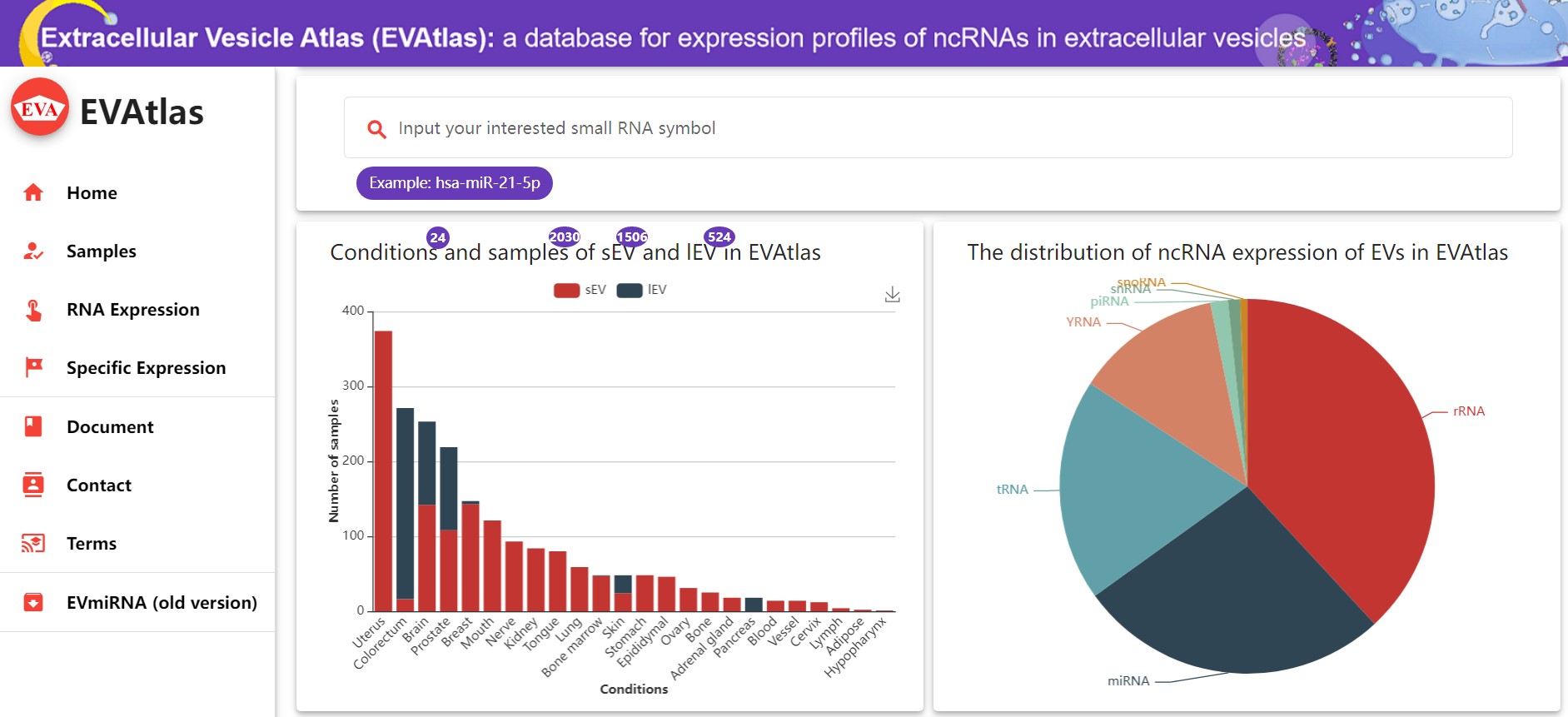
图3 EVAtlas数据库首页
EVAtlas提供了四个功能模块:(1)浏览和比较来自24种条件下8个来源(血浆、血清、唾液、尿液、精液、母乳、原代细胞和细胞系)的EV中小RNA的表达水平;(2) 根据候选ncRNA在相关组织中的表达情况对其进行优先排序;(3) 探索24种条件下 EV 中特异性表达的小RNA;(4) 浏览和查询小RNA功能、相关药物、靶基因和EV的相关分离方法。
在这里我们以“Lung”来源的EV样本为例,演示用户如何使用EVAtlas。如下图所示,用户可以从网站首页看到EVAtlas包含的模块信息(图4A)以及整个数据库中包含的数据统计(图4B)。通过点击模块并选择感兴趣的组织或EV来源(图4C),如肺(Lung),从而得到在Lung相关来源的不同疾病条件下sEV和lEV相关的研究样本以及文献(图4D)。点击感兴趣的疾病状况下的Project ID列下的紫色按钮,用户可以得到该条件下不同类型的小RNA的表达情况(图4E),以及进一步选择一个小RNA,则可得到不同组织来源的EV中该小RNA的表达趋势(图4F)。
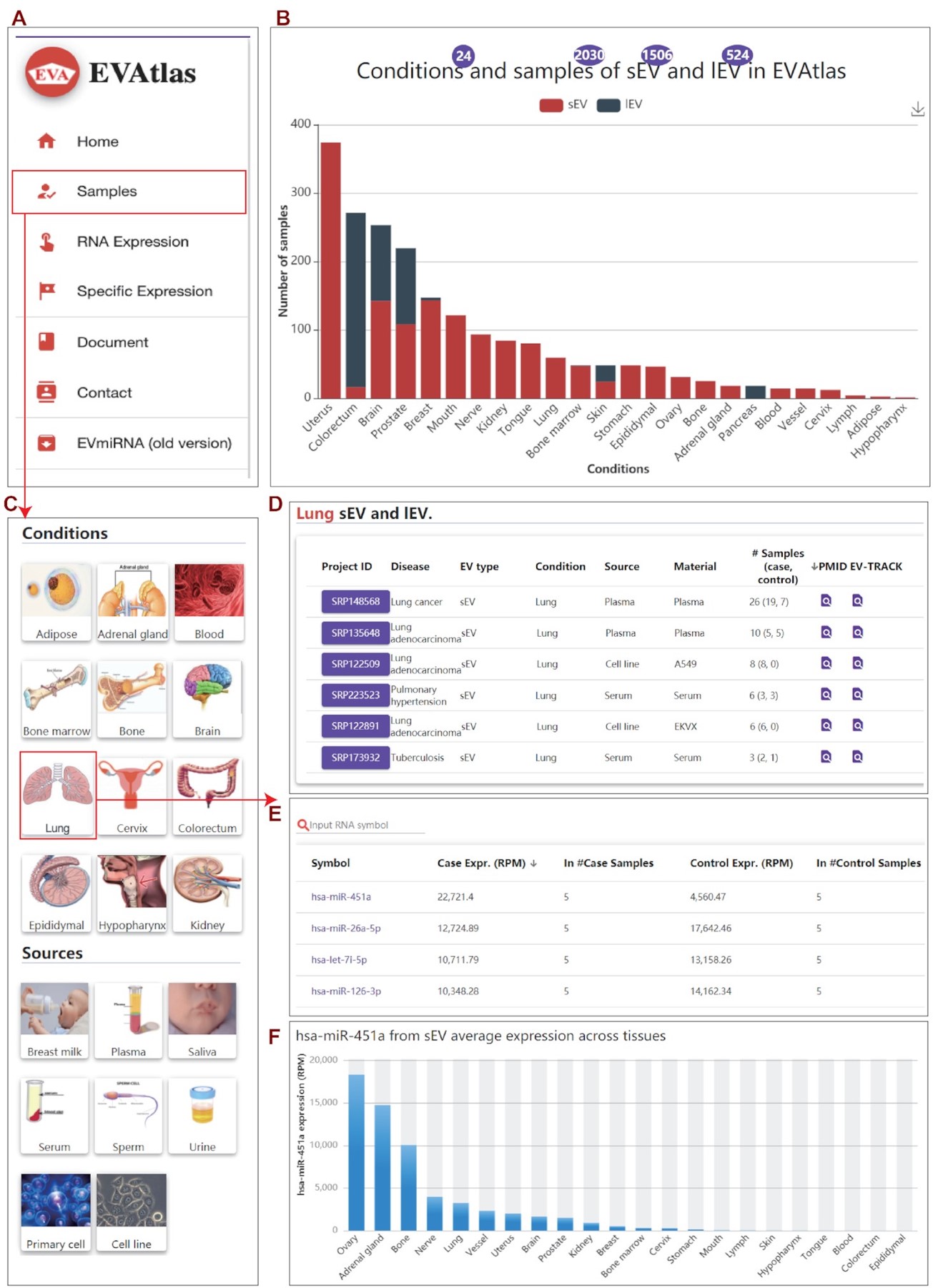
图4 EVAtlas数据库的使用示例
exRNA Atlas是细胞外 RNA(exRNA) 通讯联盟 (ERCC,英文全称) 的数据库。数据库由ERCC创立,旨在推动研究人员共享资源,建立exRNA研究的统一标准。数据库信息包括人类和小鼠生物流体中的小分子RNA测序数据和qPCR衍生的exRNA图谱,数据库使用exceRpt小RNA-seq流程处理所有小RNA-seq数据集,并且ERCC开发的质量指标被统一应用于这些数据集(如图5)。
图5 exRNA Atlas数据库首页
exoRBase是细胞外囊泡中长RNA (exLRs) 相关的数据库,这些数据来源于不同人体体液中的RNA-seq样本分析。exLRs包含信使 RNA (mRNA)、长链非编码 RNA (lncRNA) 和环状 RNA (circRNA)。
为了使数据更为丰富和准确,该数据库通过收集来自四种人体体液的约1,000个细胞外囊泡的RNA-seq数据,将exoRBase 数据库更新到了 2.0 版。exoRBase 2.0整合和可视化了RNA 表达谱,以及添加了通路水平的变化和循环细胞外囊泡起源的异质性。
exoRBase 2.0提供了exLR的全面注释和表达图谱(如图6)。它将有助于从人体体液中识别新的exLR 特征,并将有助于发现新的循环生物标志物,以改善肿瘤的诊断和治疗。
图6 exoRBase 2.0数据库首页
5. ExoBCD(https://exobcd.liumwei.org)
ExoBCD是一个全面的web数据库(https://exobcd.liumwei.org)。在该数据库中,共鉴定出3类306个有价值的EV分子,包括49个潜在生物标志物(英文全称,PBs)和257个生物感兴趣的分子(英文全称,BIM,包括121个mRNAs、172个miRNAs和13个lncRNAs),以及它们的分子特征、生物学实验、基因表达模式、总体存活率、综述文献、肿瘤分期或亚型及临床应用等详细信息(如图7)。所有这些内容被完全合并成15种注释类型和20907个条目。所有这些内容都直接或间接地来源于950篇论文、4个高质量高通量数据集的预测分析以及1191例TCGA乳腺癌病例验证,最终证明36个EV内的分子(最有价值的包括IGF1R和FRS2)成为乳腺癌预后诊断的潜在生物标志物,并提供了查询界面,方便用户浏览和可视化EV分子注释信息。总之,ExoBCD数据库整合了高通量表达数据分析和文献调研,提供了一个全面而广泛的资源,具有友好的界面和统计可视化。
图7 ExoBCD数据库首页
内容来源于《细胞外囊泡基础研究与临床应用》(陈刚主编),ISBN 978-7-03-076715-8,北京:科学出版社,2024年3月。